FastKing gDNA Dispelling RT SuperMix
အင်္ဂါရပ်များ
■မြန်ဆန်ခြင်း၊ ပုံစံ ၁၈ ခုဖြင့်သာ ၁၈ မိနစ်အတွင်းဂျီနိုအာဖယ်ရှားခြင်းနှင့်ထိရောက်သောပြောင်းပြန်စာသားဖြည့်ရန်အဆင့်တစ်ဆင့်
efficiency မြင့်မားသောထိရောက်မှု - ပြောင်းပြန် transcriptase ကို ၉၅%ထက်ကျော်လွန်သော hydrophobic motif ဖြင့်ပြုပြင်ထားသည်။
■ရိုးရှင်းပြီးလွယ်ကူသည်။ သီးသန့် thermosensitive DNase သည်မြန်ဆန်သောအကျိုးသက်ရောက်မှု၊ တိုတောင်းသောတုံ့ပြန်မှုအချိန်တိုနှင့်မြင့်မားသောထိရောက်မှုရှိပြီး cDNA ကိုမထိခိုက်ပါ။
သတ်မှတ်ချက်
အမျိုးအစား: ဗီဇပြုပြင်ထားသောပြောင်းပြန် transcriptase, gDNase
လုပ်ထုံးလုပ်နည်းများ အဆင့်တစ်ခု (မျိုးရိုးဗီဇ DNA ဖယ်ရှားခြင်းနှင့် RT)
RT ထိရောက်မှု > ၉၅%
နမူနာ 1 ng- 2 μg
စစ်ဆင်ရေးအချိန်: ~ ၁၈ မိနစ်
လျှောက်လွှာများ: ပြောင်းပြန်ကူးထားသော cDNA ကိုသမားရိုးကျ PCR, Real time PCR, cDNA စာကြည့်တိုက်တည်ဆောက်ခြင်း, SAGE (Gene Expression of Gene Expression)၊ primer extension နှင့်အခြားသမားရိုးကျစမ်းသပ်မှုများတွင်သုံးနိုင်သည်။
ထုတ်ကုန်အားလုံးသည် ODM/OEM အတွက်စိတ်ကြိုက်ပြုလုပ်နိုင်ပါသည်။ အသေးစိတ်အတွက်၊ကျေးဇူးပြုပြီး Customized Service (ODM/OEM) ကိုနှိပ်ပါ။
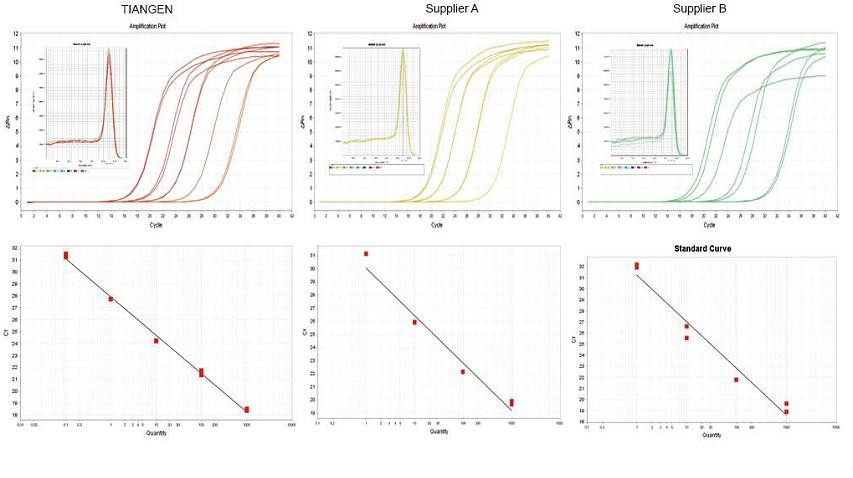 |
စမ်းသပ်ဥပမာ ၁။ cDNA ကိုထုတ်လုပ်သူ A နှင့်ရောင်း ၀ ယ် B တို့မှသက်ဆိုင်ရာထုတ်ကုန်များ၊ တစ်ဆင့်ချင်းစီပြောင်းပြန်ပမာဏ reagents များဖြင့်ပေါင်းစပ်ပြီးပေါင်းစပ်ခဲ့သည်။ TIANGEN Talent qPCR PreMix (SYBR Green) ကို သုံး၍ ကြွက်များ၏ RN5 မျိုးဗီဇကိုရှာဖွေပြီးချဲ့ခြင်းကွေးခြင်း၊ အရည်ပျော်ခြင်းကွေးညွှတ်ခြင်းနှင့်စံကွေးကောက်ခြင်းတို့ကိုခွဲခြမ်းစိတ်ဖြာခဲ့သည်။ ရလဒ်များသည် TIANGEN FastKing gDNA Dispelling RT SuperMix တွင် reverse transcription နှင့်အလွန်ကောင်းမွန်သောစိတ်ဖိစီးမှုခုခံမှုတို့အပြီးအမြင့်ဆုံးညစ်ညူးမှုအကြွင်းအကျန်များနှင့်ပုံစံခွက်များအတွက်သိသာထင်ရှားသည့်အကျိုးကျေးဇူးများရှိသည်။ |
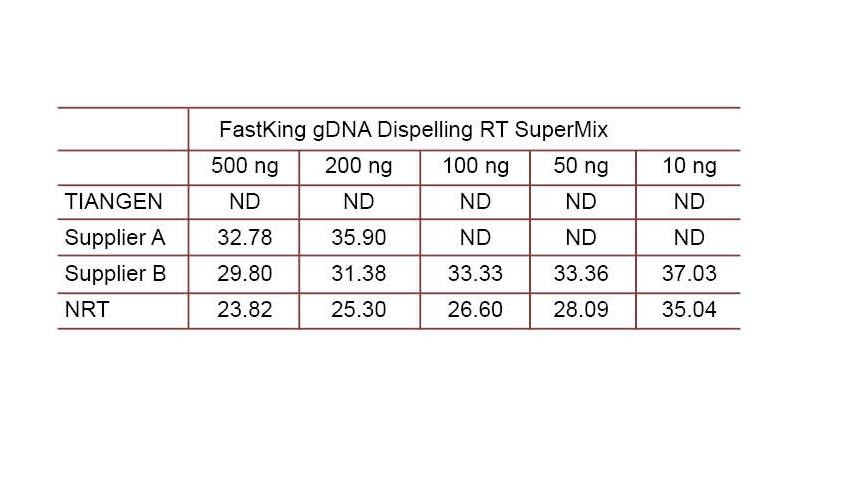 |
စမ်းသပ်ဥပမာ ၂။ cDNA ကို TIANGEN FastKing gDNA Dispelling RT SuperMix၊ ထုတ်လုပ်သူ A နှင့်ရောင်း ၀ ယ် B တို့မှသက်ဆိုင်ရာထုတ်ကုန်များဖြင့်တစ်ဆင့်ခြေလှမ်းပြောင်းပြန်ပမာဏ reagents များဖြင့်ပေါင်းစပ်ပြီးပေါင်းစပ်ခဲ့သည်။ TIANGEN Talent qPCR PreMix (SYBR Green) ကို သုံး၍ လူသား HsG မျိုးဗီဇကိုရှာဖွေပြီးကွဲပြားသောဓာတ်ကူပစ္စည်းများ၏ gDNA ဖယ်ရှားနိုင်စွမ်းကိုရှာဖွေရန် genomic DNA ကွဲပြားခြင်းကိုကိုယ်တိုင်ထည့်ပါ။ Ct ရလဒ်များက TIANGEN FastKing gDNA Dispelling RT SuperMix သည်မျိုးရိုးဗီဇ DNA ကိုဖယ်ရှားနိုင်သောအစွမ်းရှိသည်။ မျိုးရိုးဗီဇ DNA အကြွင်းအကျန် ၅၀၀ ng အထိကိုရလဒ်မထိခိုက်ဘဲစုံလင်စွာဖယ်ရှားနိုင်သည်။ ND: မတွေ့ပါ။ NRT: ပြောင်းပြန်စာသားမပါဘဲရောနှောခြင်းကိုရှာဖွေခြင်း။ |
A-1 RNA သည်ပျက်စီးသွားသည်
- ညစ်ညမ်းမှုမရှိသောအရည်အသွေးမြင့် RNA ကိုသန့်စင်ပါ။ RNA မှထုတ်ယူထားသောပစ္စည်းသည် RNA ပျက်စီးခြင်းကိုကာကွယ်ရန်အတတ်နိုင်ဆုံးလတ်ဆတ်သင့်သည်။ RT တုံ့ပြန်မှုမတိုင်မီ denatured gel တွင် RNA သမာဓိကိုခွဲခြမ်းစိတ်ဖြာပါ။ RNA ထုတ်ယူပြီးနောက်၎င်းကို ၁၀၀% formamide ဖြင့်သိမ်းဆည်းသင့်သည်။ RNase inhibitor ကိုသုံးလျှင်အပူအပူချိန် <၄၅ ဒီဂရီစင်တီဂရိတ်ရှိသင့်ပြီး pH သည် ၈.၀ ထက်နည်းသင့်သည်။ ထို့ပြင် RNase inhibitor ကို≥ 0.8 mM DTT ပါ ၀ င်သော solution များတွင်ထည့်သင့်သည်။
A-2 RNA တွင် reverse transcription reaction ၏ inhibitors များပါ ၀ င်သည်
———————————————————————————————————————————————————————————————————————————————————————————————————————————————————————————————————————— SDS, EDTA, glycerol, sodium pyrophosphate, spermidine, formamide, guanidine salt, etc စသည်ဖြင့်ထိန်းချုပ်မှု RNA ကိုနမူနာနှင့်ရောနှောပြီးအထွက်ထိန်းချုပ်မှုရှိမရှိစစ်ဆေးရန်ထိန်းချုပ် RNA တုံ့ပြန်မှုနှင့်နှိုင်းယှဉ်ပါ။ inhibitors များကိုဖယ်ရှားရန် RNA မိုးရွာသွန်းမှုကို ၇၀% (v/v) ethanol ဖြင့်ဆေးပါ။
A-3 ပထမ ဦး ဆုံးသော cDNA ကိုပေါင်းစပ်ရန်သုံးသော primer များကိုလုံလောက်စွာမီးမလုံပါ
—— စမ်းသပ်ခြင်းတွင်သုံးသော primers များအတွက်သင့်လျှော်သောအပူချိန်သည်သင့်တော်ကြောင်းဆုံးဖြတ်ပါ။ အမှတ်တမဲ့ hexamers များအတွက်တုံ့ပြန်မှုအပူချိန်မရောက်မီ ၁၀ မိနစ် ၂၅ ဒီဂရီစင်တီဂရိတ်တွင်အပူချိန်ထိန်းထားရန်အကြံပြုသည်။ မျိုးရိုးအလိုက်သီးသန့် primers (GSP) အတွက်အခြား GSP ကိုစမ်းပါ၊ သို့မဟုတ် oligo (dT) သို့မဟုတ် random hexamer သို့ပြောင်းပါ။
A-4 RNA စတင်ခြင်းပမာဏအနည်းငယ်
- RNA ပမာဏကိုတိုးပါ။ ၅၀ ng ထက်နည်းသော RNA နမူနာများအတွက် 0.1 μgမှ 0.5 μg acetyl BSA ကိုပထမ ဦး ဆုံး cDNA ပေါင်းစပ်မှုတွင်သုံးနိုင်သည်။
A-5 ပစ်မှတ်အစီအစဉ်ကိုခွဲခြမ်းစိတ်ဖြာထားသောတစ်သျှူးများတွင်ဖော်ပြမထားပါ။
--- အခြားတစ်သျှူးများကိုစမ်းကြည့်ပါ။
A-6 PCR တုံ့ပြန်မှုမအောင်မြင်ပါ
—— အဆင့်နှစ်ဆင့်ရှိသော RT-PCR များအတွက် PCR အဆင့်ရှိ cDNA ပုံစံသည်တုံ့ပြန်မှုပမာဏ၏ ၁/၅ ထက်မပိုနိုင်ပါ။
A-1 primer များနှင့်တင်းပလိတ်များကိုမတိကျသောမီးညှိခြင်း
primers ၏ 3'-end တွင် 2-3 dG သို့မဟုတ် dC မပါသင့်ပါ။ Random primers (သို့) oligo (dT) အစားပထမဆုံး strand ပေါင်းစပ်မှုတွင် Gene အတွက်သီးသန့် primers ကိုသုံးပါ။ ပထမသံသရာအနည်းငယ်တွင်ပိုမိုမြင့်မားသော annealing အပူချိန်ကိုသုံးပါ၊ ထို့နောက် annealing အပူချိန်ကိုလျှော့ပါ။ တုံ့ပြန်မှု၏တိကျမှုကိုတိုးတက်စေရန် PCR အတွက် hot-start Taq DNA polymerase ကိုသုံးပါ။
A-2 သည်မျိုးရိုးအလိုက်တိကျသော primers များ၏ဒီဇိုင်းညံ့ဖျင်းသည်
--- အသံချဲ့စက် primer ဒီဇိုင်းအတွက်တူညီသောအခြေခံအချက်များအတိုင်းလိုက်နာပါ။
A-3 RNA သည် genomic DNA နှင့်ညစ်ညမ်းသည်
DNA ညစ်ညမ်းမှုကိုရှာဖွေရန်ပြောင်းပြန်စာသားမပါဘဲထိန်းချုပ်မှုတုံ့ပြန်မှုတစ်ခုသတ်မှတ်ပါ။
A-4 primer dimer ကိုဖွဲ့စည်းခြင်း
၃- အဆုံးတွင်အပိုအတွဲများမပါသောဒီဇိုင်းများ
A-5 မြင့်လွန်းသော Mg၂+ အာရုံစူးစိုက်မှု
- Mg ကိုပိုကောင်းအောင်လုပ်ပါ၂+ ပုံစံတစ်ခုစီနှင့် primer ပေါင်းစပ်မှုအတွက်အာရုံစူးစိုက်မှု
A-6 သည်နိုင်ငံခြား DNA နှင့်ညစ်ညမ်းသည်
—— aerosol ခံနိုင်ရည်ရှိသောအကြံပေးချက်များနှင့် UDG အင်ဇိုင်းများကိုသုံးပါ။
A-1 ပထမကြိုး၏ထုတ်ကုန်၏ပါဝင်မှုသည်မြင့်မားသည်
သမားရိုးကျ PCR တုံ့ပြန်မှုအဆင့်တွင်ပထမ ဦး ဆုံးသောကုန်ပစ္စည်း၏ပမာဏကိုလျှော့ချပါ။
A-2 PCR တုံ့ပြန်မှုတွင် primer ပမာဏမြင့်မားသည်
--- primer ထည့်သွင်းမှုကိုလျှော့ချပါ။
A-3 သံသရာများလွန်းသည်
—— PCR တုံ့ပြန်မှုအခြေအနေများကိုမြှင့် တင်၍ PCR စက်ဝန်းအရေအတွက်ကိုလျှော့ချပါ။
A-4 အပူပေးစနစ်အလွန်နည်းသည်
—- သတ်မှတ်ထားသောစတင်ခြင်းနှင့်တိုးချဲ့ခြင်းကိုကာကွယ်ရန် annealing အပူချိန်ကိုတိုးပါ။
A-5 သည် DNA ညစ်ညမ်းမှုကိုကာကွယ်ရန်အရည်အသွေးမြင့် RNA ကိုထုတ်ယူသော oligonucleotide အပိုင်းအစများမဟုတ်သောသီးခြားအသံချဲ့ခြင်း။
RT-PCR သည် RNA ကို cDNA သို့ပြောင်းပြန်ပြောင်းရန်နှင့်၎င်းအားပစ်မှတ်အပိုင်းအစကိုချဲ့ထွင်ရန် PCR တုံ့ပြန်မှုအတွက်ပုံစံခွက်အဖြစ်သုံးသည်။ စမ်းသပ်မှု၏သီးခြားအခြေအနေများအရ random primers, Oligo dT နှင့် gene specific primers များကိုရွေးချယ်ပါ။ အထက်ပါ primer အားလုံးကို hairpin structure မပါဘဲ eukaryotic cell mRNA တိုအတွက်သုံးနိုင်သည်။
ကျပန်း primer: ဆံထိုးဖွဲ့စည်းပုံပါသော RNA ရှည်များအပြင် rRNA, mRNA, tRNA စသဖြင့် RNA အမျိုးအစားအားလုံးအတွက်၎င်းတို့ကိုအများအားဖြင့်ပုံစံခွက်တစ်ခု၏ RT-PCR တုံ့ပြန်မှုအတွက်သုံးသည်။
Oligo dT: PolyA tailing ပါ ၀ င်သော RNA အတွက်သင့်တော်သည် (prokaryotic RNA, eukaryotic Oligo dT rRNA နှင့် tRNA တွင် PolyA tails မရှိ) Oligo dT သည် PolyA အမြီးနှင့်ချည်နှောင်ထားသောကြောင့် RNA နမူနာများ၏အရည်အသွေးသည်မြင့်ရန်လိုအပ်ပြီးပျက်စီးခြင်းပမာဏအနည်းငယ်သည်ပင်အစအဆုံး cDNA ပေါင်းစပ်မှုပမာဏကိုများစွာလျော့ကျစေလိမ့်မည်။
မျိုးရိုးအလိုက်သီးသန့် primer
နည်းလမ်းနှစ်ခုရှိပါတယ်။
၁။ ပြည်တွင်းရည်ညွှန်းနည်းလမ်း - သီအိုရီအရ cDNA သည်ကွဲပြားသောအရှည်များရှိ DNA အပိုင်းအစများဖြစ်သောကြောင့် electrophoresis ၏ရလဒ်သည်လိမ်းကျံသည်။ RNA ကြွယ်ဝမှုနိမ့်လျှင် electrophoresis တွင်မည်သည့်ထုတ်ကုန်မျှပြလိမ့်မည်မဟုတ်သော်လည်း PCR ဖြင့်မည်သည့်ထုတ်ကုန်ကိုမျှချဲ့ထွင်လိမ့်မည်ဟုမဆိုလိုပါ။ ယေဘုယျအားဖြင့် cDNA ကိုရှာဖွေရန်ပြည်တွင်းရည်ညွှန်းချက်ကိုသုံးနိုင်သည်။ ပြည်တွင်းရည်ညွှန်းချက်ရလဒ်များရရှိပါက cDNA ၏အရည်အသွေးကိုအခြေခံအားဖြင့်အာမခံနိုင်သည် (အချို့ကိစ္စများတွင်ပန်းတိုင်ဗီဇအပိုင်းအစရှည်လွန်းလျှင်ခြွင်းချက်များရှိနိုင်သည်) ။
၂။ ဤပုံစံဖြင့်ချဲ့ထားသောလူသိများသောမျိုးဗီဇရှိလျှင်၎င်းကိုဤမျိုးဗီဇ၏အခြေခံများဖြင့်အတည်ပြုနိုင်သည်။ ပြည်တွင်းရည်ညွှန်းချက်ချဲ့ခြင်းသည် cDNA နှင့် ပတ်သက်၍ ပြဿနာမရှိဟုမဆိုလိုပါ။ ပြည်တွင်းရည်ညွှန်းချက်၌ cDNA ၌ကြွယ်ဝမှုမြင့်မားသောကြောင့်၎င်းကိုချဲ့ထွင်ရန်လွယ်ကူသည်။ အကြောင်းအမျိုးမျိုးကြောင့် cDNA သည်တစ်စိတ်တစ်ပိုင်းပျက်စီးသွားပါကဖြစ်နိုင်ခြေရှုထောင့်မှကြည့်လျှင် PCR နည်းပါးသောပစ်မှတ်မျိုးဗီဇရလဒ်များသည်များစွာထိခိုက်လိမ့်မည်။ ပြည်တွင်းရည်ညွှန်းချက်သည်များပြားနေဆဲဖြစ်သော်လည်းအသံချဲ့စက်သည်ထိခိုက်လိမ့်မည်မဟုတ်ပေ။
RNA ၏တစ်စိတ်တစ်ပိုင်းကိုကျဆင်းစေသည်။ ခိုင်မာမှုကိုရှာဖွေပြီး RNA ကိုသန့်စင်ပါ
ကွဲပြားသောမျိုးစိတ်များ၏ RNA ပါဝင်မှုကွဲပြားနိုင်သည်၊ သို့သော်ယေဘုယျအားဖြင့်ထုတ်ယူထားသောစုစုပေါင်း RNA တွင် gel electrophoresis တွင်ကြည်လင်သော 28S နှင့် 18S တီးဝိုင်းနှစ်ခုပါ ၀ င်သင့်သည်။ 5S band သည် RNA ပျက်စီးသွားပြီဖြစ်ကြောင်းနှင့်၎င်း၏တောက်ပမှုသည်ပျက်စီးခြင်းအတိုင်းအတာနှင့်အချိုးကျသည်။ ပြည်တွင်းရည်ညွှန်းချက်ကိုအောင်မြင်အောင်ချဲ့ခြင်းသည် RNA နှင့် ပတ်သက်၍ ပြသနာမရှိဟုမဆိုလိုပါ၊ အတွင်းပိုင်းရည်ညွှန်းချက်သည်မြင့်မား နေ၍ ပျက်စီးခြင်းသည်မပြင်းထန်သရွေ့ RNA ကိုချဲ့နိုင်သည်။ OD သည်260/OD280spectrophotometer ဖြင့်တိုင်းတာသောစင်ကြယ်သော RNA အချိုးသည် ၁.၉ နှင့် ၂.၁ အကြားဖြစ်သင့်သည်။ RNA တွင်ပရိုတင်းအနည်းငယ်ညစ်ညမ်းမှုသည်အချိုးကိုလျှော့ချလိမ့်မည်။ တန်ဖိုးကမနိမ့်သရွေ့ RT ကထိခိုက်မှာမဟုတ်ဘူး။ RT အတွက်အရေးအကြီးဆုံးအရာမှာ RNA သမာဓိဖြစ်သည်။
ပြည်တွင်းရည်ညွှန်းဗီဇ၏တိုးချဲ့မှုသည် RT အောင်မြင်ကြောင်းကိုသာညွှန်ပြနိုင်သော်လည်း၎င်းသည် cDNA strand ၏အရည်အသွေးနှင့်မသက်ဆိုင်ပါ။ အတွင်းပိုင်းရည်ညွှန်းချက်အပိုင်းအစများသည်ယေဘုယျအားဖြင့်သေးငယ်ပြီးထုတ်ဖော်ပြသမှုမြင့်မားသောကြောင့်၎င်းတို့ကိုပြောင်းပြန်စာကူးရာမှာအောင်မြင်ဖို့ပိုလွယ်တယ်။ သို့သော်ပစ်မှတ်ဗီဇ၏အရွယ်အစားနှင့်မျိုးဗီဇသည်ဗီဇမှဗီဇကွဲပြားသည်။ cDNA အရည်အသွေးကိုအထူးသဖြင့် 2 kb ထက်ပိုသောပစ်မှတ်အပိုင်းအစများအတွက်ပြည်တွင်းရည်ညွှန်းချက်ဖြင့်သာဆုံးဖြတ်နိုင်သည်။
အချို့သောနမူနာများသည်ရှုပ်ထွေးသောအလယ်အလတ်ဖွဲ့စည်းပုံများရှိသည်၊ သို့မဟုတ်ကြွယ်ဝသော GC ပါဝင်မှုရှိသည် (သို့) ကြွယ်ဝမှုနည်းပါးခြင်းဖြင့်အဖိုးတန်သည်။ ဤအခြေအနေများတွင်သင့်လျော်သော reverse transcriptase ကိုပစ်မှတ်အပိုင်းအစနှင့်နမူနာအရအရွယ်အစားအလိုက်ရွေးချယ်သင့်သည်။ မြင့်မားသော GC ပါဝင်မှုများနှင့်ရှုပ်ထွေးသောဒုတိယဖွဲ့စည်းပုံပါ ၀ င်သော RNA တင်းပလိတ်များအတွက်အလယ်အလတ်ပုံစံကိုအပူချိန်နိမ့်ခြင်း (သို့) ပုံမှန် reverse transcriptase ဖြင့်ဖွင့်ရန်ခက်ခဲသည်။ ဤတမ်းပလိတ်များအတွက် Quant Reverse Transcriptase ကိုရွေးချယ်နိုင်သည်၊ အကြောင်းမှာ၎င်း၏ reverse transcription လုပ်ဆောင်ချက်သည် MNA MLV series reverse transcriptase ထက်သိသာထင်ရှားသည်။ ၎င်းသည် RNA အမျိုးမျိုးကိုစာသားများကိုထိထိရောက်ရောက်ကူးပြောင်းနိုင်ပြီး RNA ကို cnc ရဲ့ပထမ strand သို့အများဆုံးအတိုင်းအတာအထိကူးပြောင်းပေးနိုင်သည်။ ယေဘူယျ reverse transcriptase kit ကိုသုံးသောအခါ 20 systeml system သည်စုစုပေါင်း RNA ၏ 1 μgကိုသာထိရောက်စွာမှတ်တမ်းတင်နိုင်သည်။ ကိရိယာ၏အများဆုံး RT စွမ်းရည်ကိုအာရုံစိုက်ပါ။ ပုံစံခွက်ကိုပိုထည့်လျှင်၊ ပြောင်းပြန်စာသားမှတ်တမ်းသည် RNA ကိုကြွယ်ဝမှုမြင့်မားစေလိမ့်မည်။ ထို့ကြောင့်စနစ်၏အမြင့်ဆုံးစွမ်းရည်ထက်မကျော်လွန်ခြင်းသည်ကောင်း၏။
A-1 သည် RNA ကိုဆိုးရွားစွာပျက်စီးစေသလား၊ RT အောင်မြင်လျှင်ဖြစ်စေဆုံးဖြတ်ပါ
ယေဘုယျအားဖြင့်ပြည်တွင်းရည်ညွှန်းချဲ့ထွင်မှုမအောင်မြင်ရခြင်းအကြောင်းအရင်းသည်ပြင်းထန်သော RNA ပျက်စီးခြင်းကြောင့်ဖြစ်လေ့ရှိသည်။ နောက်ထပ်ဖြစ်နိုင်သောအကြောင်းအရင်းမှာ reverse transcription ပျက်ကွက်ခြင်းဖြစ်သည်။ RD ၏အရည်အသွေးပြဿနာမရှိလျှင်ပြောင်းပြန်လှန်ကူးယူမှုအောင်မြင်သလားဆုံးဖြတ်ရန်စံအဖြစ်သုံးနိုင်သည်။ ပြောင်းပြန်ကူးယူခြင်းလုပ်ငန်းစဉ်၌အရေးအကြီးဆုံးအရာမှာတုံ့ပြန်မှုထိရောက်မှုကိုတိုးတက်စေရန်အမြဲမပြတ်အပူချိန်နှင့်စဉ်ဆက်မပြတ်တုံ့ပြန်မှုစနစ်ကိုထိန်းသိမ်းရန်ဖြစ်သည်။
A-2 အတွင်းပိုင်းရည်ညွှန်းဗီဇများချဲ့ခြင်းအတွက် primers များသည်ယုံကြည်စိတ်ချရမှုရှိမရှိနှင့် PCR တွင်အသုံးပြုသောဓာတ်ကူပစ္စည်းများနှင့် ပတ်သက်၍ ပြဿနာတစ်စုံတစ်ရာရှိလျှင်ဆုံးဖြတ်ပါ။
နှိုင်းရပမာဏအတွက် RNA ကို reverse transcription မတိုင်မီအရေအတွက်တွက်ရမည်၊ ဥပမာ reverse reverse transcription kit များစွာတွင်လိုအပ်သောဥပမာအားဖြင့် RNA input ကို 1 μgအဖြစ်တွက်ချက်ပါ။ ပြောင်းပြန်ကူးထားသော cDNA သည် RNA, oligo dT, enzyme, dNTP နှင့် DNA အနည်းငယ်တို့ပင်ရောနှောနေသောကြောင့်သွေဖည်ခြင်းကိုဖြစ်ပေါ်စေသောကြောင့် cDNA ကိုအတိအကျတွက်ရန်မဖြစ်နိုင်ပေ။ ထို့ကြောင့် RNA ပမာဏတွက်ချက်ရန်လိုအပ်သည်။ ပြောင်းပြန်ကူးယူခြင်း၏ထိရောက်မှုသည်မတူညီသောနမူနာများအကြားတူညီသည်ဟုယူဆခြင်း၊ ရရှိသော cDNA ပမာဏသည်တူညီသင့်သည်၊ အရေအတွက်ခွဲခြမ်းစိတ်ဖြာခြင်းသည်စုစုပေါင်း RNA ၏တူညီသောမျိုးရိုးဗီဇများ၏ကွဲပြားခြားနားသောမျိုးဗီဇများ၏နှိုင်းယှဉ်မှုကိုပြနိုင်သည်။ နှိုင်းယှဉ် fluorescence quantitative PCR ကိုလုပ်ဆောင်သည့်အခါ internal transcation ကို reference အဖြစ်ဆောင်ရွက်နိုင်သောကြောင့် quantitative cDNA သည်မလိုအပ်နိုင်ပါ။
၎င်းသည်အဓိကအားဖြင့်မျိုးရိုးဗီဇများနှင့်ဆက်စပ်နေပြီးရှည်လျားသောအပိုင်းအစများကိုပြောင်းပြန်ကူးခြင်းသည်မျိုးဗီဇအများစုအတွက်မဖြစ်နိုင်ပါ။ ပထမအချက်မှာ reverse transcription ၏ထိရောက်မှုသည် PCR ထက်များစွာနိမ့်သည်။ ဒုတိယအချက်မှာ GC ကြွယ်ဝသောဒေသနှင့်မျိုးဗီဇများစွာ၏ဆင့်ပွားဖွဲ့စည်းပုံသည်ပြောင်းပြန်ကူးယူခြင်းနှင့် PCR နှစ်ခုစလုံးကိုကန့်သတ်ထားသည်။ နောက်ဆုံးတွင် PCR ၏သစ္စာရှိမှုနှင့်ချဲ့ထွင်မှုထိရောက်မှုသည်တစ်ချိန်တည်းတွင်အာမခံရန်ခက်ခဲသည်။ ပြောင်းပြန်ကူးယူခြင်းလုပ်ငန်းစဉ်တွင်၊ အထူးသဖြင့် oligo dT ကို အသုံးပြု၍ အနိမ့်မိတ္တဗီဇအတွက်ရှည်လျားသောအပိုင်းအစကိုမည်သူမျှအာမခံနိုင်မည်မဟုတ်ပေ။ ပိုများသော GC ရှိသော 5 'UTR အတွက်၎င်းသည် ပို၍ ပင်ခက်ခဲသည်။ ထို့ကြောင့်၎င်းသည်အမှတ်အသားများကိုကျပန်း primers များဖြင့်ပြောင်းပြန်ရေးရန်၊ ပစ်မှတ်အပိုင်းအစ၌သဘာဝကွဲအက်နေသောနေရာများကိုရှာဖွေ။ အပိုင်းများဖြင့်ချဲ့ထွင်ခြင်း၊ ထို့နောက်ကန့်သတ်ချက်အစာခြေခြင်းနှင့် ligation တို့ကိုလုပ်ဆောင်ရန်ကျိုးကြောင်းဆီလျော်သောနည်းလမ်းတစ်ခုဖြစ်နေသေးသည်။ ယေဘူယျအားဖြင့်၎င်းသည် 2 kb ထက်ပိုကြီးသောအပိုင်းအစများကိုတိုက်ရိုက်ချဲ့ရန်ခက်ခဲသော်လည်း၎င်းကိုအမြဲရယူရန်မဖြစ်နိုင်ပေ။ ၁။ ပထမ ဦး စွာ RNA/mRNA ၏ခိုင်မာမှုကိုအာမခံ။ TRIZOL ထုတ်ယူခြင်းကို ဦး စားပေးသည်။ 2.M-MLV RT-PCR kit ကိုတိုက်ရိုက်သုံးနိုင်သည်။ မီးပိတ်သောအချိန်ကိုတိုးချဲ့။ ချဲ့ခြင်းလုပ်ငန်းစဉ်၌စက်ဝန်းနံပါတ်ကိုစနစ်တကျတိုးပါ။ တနည်းအားဖြင့် nested PCR ကိုသုံးနိုင်သည်၊ သို့မဟုတ်အစိတ်အပိုင်းများကိုပုံမှန် PCR ချဲ့ထွင်မှုမတိုင်မီသင့်လျော်သောတိုးချဲ့ denaturation နှင့်တိုးချဲ့ချိန်တို့ဖြင့်ပထမသို့မဟုတ်နှစ်ကြိမ်တုံ့ပြန်နိုင်သည်။ polymerase ၏သစ္စာရှိမှုကိုဂရုပြုပါ။ 3. Long Taq ကိုစံရလဒ်များရရှိရန် PCR တွင်သုံးနိုင်သည်။ ၄။ ပရိုတိန်းထုတ်ဖော်ပြသခြင်းအတွက် high fidelity polymerase ကိုသုံးသင့်သည်။
TIANGEN မှကမ်းလှမ်းသော reverse transcriptase နှစ်မျိုးရှိသည်။ Quant/King RTase နှင့် TIANScript M-MLV ၎င်းတို့အကြားအဓိကကွာခြားချက်သည်တင်းပလိတ်များထည့်သွင်းမှုပမာဏဖြစ်သည်။ Quant သည်ထူးခြားသော reverse transcriptase တစ်ခုဖြစ်ပြီး Moloney murine leukemia virus မှဆင်းသက်လာသောအသုံးများသော M-MLV နှင့်ကွဲပြားသည်။ Quant သည်အင်ဂျင်နီယာ Escherichia coli မှ recombinantly recombinantly recombinantly ထုတ်ဖော်ပြောကြားသောအသစ်သောစွမ်းဆောင်ရည်မြင့် reverse transcriptase အသစ်တစ်ခုဖြစ်သည်။ Quant သည် 50 ng-2 μg RNA ကိုမြင့်မားသောပြောင်းပြန်အသံသွင်းခြင်းလုပ်ဆောင်ချက်နှင့်အထွက်နှုန်းမြင့်မားရန်သင့်တော်သည်။ သာမန် MMLV (သို့) AMV နှင့်နှိုင်းယှဉ်လျှင် Quant ၏အကြီးမားဆုံးသောလက္ခဏာမှာ၎င်းသည် RNA တင်းပလိတ်များနှင့်အလွန်ခိုင်ခံ့မှုရှိပြီးအပူချိန်မြင့်မားမှုကို denaturation မရှိဘဲစာသားမှတ်တမ်းများကိုပြောင်းပြန်လှန်နိုင်သည်။ ပိုမိုမြင့်မားသော GC ပါဝင်သောတင်းပလိတ်များအတွက်ပြောင်းပြန်လုပ်ရည်ကိုင်ရည်သည်ပိုမိုမြင့်မားသည်။ သို့သော်ဤ reverse transcriptase တွင် RNase H လုပ်ဆောင်ချက်ပါ ၀ င်သည် (၄.၅ kb တင်းပလိတ်များအတွက်သင့်တော်သည်) ။ သမားရိုးကျပြောင်းပြန်စာသားအတွက် TIANScript MMLV reverse transcriptase ကိုအကြံပြုသည်။ ဤ RTase သည်အလွန်အားနည်းသော RNase H လုပ်ဆောင်ချက်ပါ ၀ င်သောပြုပြင်ထားသောအင်ဇိုင်းတစ်ခုဖြစ်သည်၊ ၎င်းသည်ရှည်လျားသော (> 5 kb) cDNA ပေါင်းစပ်မှုအတွက်သင့်တော်သည်။
အဆင့်တစ်ဆင့်ပြောင်းပြန်ကူးယူခြင်းနှင့် PCR ချဲ့ခြင်းတို့သည်ညစ်ညမ်းမှုကိုလျှော့ချရန်အထောက်အကူဖြစ်သော cDNA ပေါင်းစပ်ခြင်းနှင့်ချဲ့ခြင်းအကြားပြွန်ကိုဖွင့်စရာမလိုဘဲတစ်ခုတည်းသောပြွန်၌ပြီးစီးသည်။ ရရှိသော cDNA နမူနာအားလုံးကိုအသံချဲ့စက်အတွက်သုံးသောကြောင့်အာရုံခံနိုင်စွမ်းသည်အနည်းဆုံး RNA စုစုပေါင်း၏ ၀.၀၁ pg ထက်ပိုမြင့်သည်။ အောင်မြင်သောခြေလှမ်း RTPCR တစ်ခုအတွက် gen-specific primers များအားယေဘူယျအားဖြင့် cDNA ပေါင်းစပ်မှုစတင်ရန်သုံးသည်။ နှစ်ဆင့်ဆင့်နည်းလမ်းဟုခေါ်သော reverse transcription နှင့် PCR amplification ကိုအဆင့်နှစ်ဆင့်ဖြင့်ဆောင်ရွက်သည်။ ပထမ ဦး စွာပြောင်းပြန်စာသားကို cDNA ရယူရန် RNA template တစ်ခုမှလုပ်ဆောင်ပြီးရရှိသော cDNA သည်တစ်ခုသို့မဟုတ်တစ်ခုထက်ပိုသောကွဲပြားသော PCR တုံ့ပြန်မှုများနှင့်ကြုံရသည်။ အဆင့်နှစ်ဆင့်ပါနည်းလမ်းသည် oligo (dT) သို့မဟုတ် random primers ကို သုံး၍ cDNA ၏ပထမ ဦး ဆုံးသောကြိုးကိုပေါင်းစပ်ရန်လမ်းညွှန်ပေးပြီး mRNA အချက်အလက်အားလုံးကိုသတ်သတ်မှတ်မှတ်နမူနာတစ်ခုမှကူးပြောင်းနိုင်သည်။
ထုတ်ကုန်အမျိုးအစားများ
အမေရိကန်ကိုဘာကြောင့်ရွေးတာလဲ
စတင်တည်ထောင်ကတည်းကကျွန်ုပ်တို့၏စက်ရုံသည်နိယာမကိုလိုက်နာခြင်းဖြင့်ပထမကမ္ဘာအဆင့်ထုတ်ကုန်များကိုတီထွင်ခဲ့သည်
အရည်အသွေးပထမ ကျွန်ုပ်တို့၏ထုတ်ကုန်များသည်စက်မှုလုပ်ငန်းတွင်အလွန်နာမည်ကောင်းရခဲ့ပြီးဖောက်သည်အသစ်နှင့်အဟောင်းများကြားတွင်အဖိုးတန်လက်ဖွဲ့ပစ္စည်းများဖြစ်သည်။